通过突变两个Cas9核酸酶域中的一个,研究人员创造了CRISPR-nickase。Nickas产生单链而不是双链断裂,当与两个相邻的GRNA一起使用时,可以降低偏离目标编辑的概率。在这篇文章中,我们将总结IDT(集成DNA技术)如何首次证明CRISPR Nickas是如何改进的同源定向修复价格,并分享他们为你的下一次CRISPR nickase实验设计规则
Cas9-nickase综述
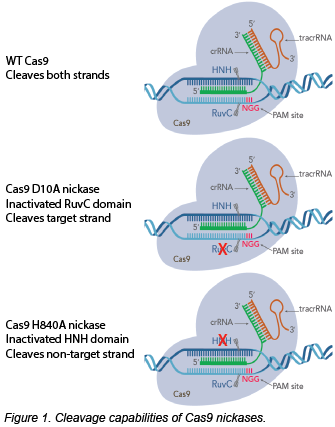
在这篇文章中,我们将使用SpCas9 nickases作为例子。D10A突变使RuvC结构域失活,因此这个nickase只切割目标链。相反,HNH结构域的H840A突变会产生一个非目标链切割镍酶。不用WT Cas9和一个gRNA直接切割两条链,你可以使用一个Cas9 nickase和两个gRNA创建一个交错切割。
对于nickase应用程序,一个常见的问题是:GRNA应该如何相互定位?gRNAs必须针对不同的链来创建DSB,但这可以通过PAM in或PAM out定向来实现。正如名称所暗示的,PAM OUT设计在目标区域的极端具有PAM序列,而PAM在设计中将目标放置在目标区域的中间,如下图所示。

为了确定最佳nickase设计,IDT科学家Mollie Schubert和Shuqi Yan设计了实验,用HEK293细胞测试nickase偏好。他们首先比较了D10A和H840A在PAM in和PAM out配置中不同刻痕距离(40-130 nt)的总编辑效率。从这些结果可以明显看出,PAM out配置中的编辑要高得多。此外,D10A的编辑效率始终高于H840A,尤其是在切口距离较小的情况下。使用D10A,他们随后表明,当GRNA太近(7-23 nt缺口距离)时,编辑效率非常低。
D10A和H840A都是强大的编辑器,但它们的indel配置文件各不相同。在PAM out配置中,D10A倾向于生成小的删除,而H840A倾向于大的插入。这些剖面可能是由于Nickas产生的不同悬挑模式造成的;D10A在PAM out设计中创建5'悬挑,H840A创建3'悬挑。
探索镍基同源定向修复
使用Nickas治疗癌症的潜在益处HDR目标范围:使用单个gRNA和WT Cas9,修复水平从切口处迅速下降10 bp.所以如果你不能找到一个好的gRNA在靠近插入位点的地方,你就不能获得高的HDR效率。尼克斯造成了交错的割伤——这个系统能在尼克斯之间的整个区域进行修复吗?为了找到答案,Schubert和Yan设计了一个pamout nickase实验,使用单链寡核苷酸(ssODN)供体插入EcoRI位点。使用D10A镍基酶在镍基之间的不同位置插入一个EcoRI位点,他们观察到>20%的修复(最大27%),通过使用具有40 bp同源臂的上链或下链ssODN供体在51 nt区域。HDR的效率优于使用WT Cas9与左侧gRNA穿过窗口。在随后的实验中,他们也能够使用具有100 nt同源臂的IDT Megamer长ssDNA引入更长的插入(mCherry)。

舒伯特和杨接着研究了一种被认为不适合HDR的情况。在AAVS1而离靶位点最近的两个grna的切割位点距离靶位点为12 nt和13 nt,而不是最佳的<10 nt距离。利用两个离靶点较远的gRNA位点,他们设计了一种D10A nickase策略(46 nt nick distance, pamout),看看能否解决这个问题。D10A nickase在很大程度上优于两个WT Cas9位点,表现出>20%的修复效率。显然,镍酶策略可以扩大靶向区域,这在没有接近预期突变位点的有效引导时尤其有用。

nickase设计的快速提示
准备好为下一个CRISPR实验使用Nickas了吗?以下是IDT的最佳建议:
- 使用PAM out配置
- 优化你的间距
- D10A:划痕部位间隔37- 68bp
- H840A:缺口部位被51-68 bp隔开
- 将D10A用于HDR
- 将预期插入件放置在缺口位置之间
- 对小的插入或标签使用40 nt同源臂(ssODN;IDT片剂®寡核苷酸)
- 对于大的插入(长ssDNA;IDT Megamer®ssDNA片段)
- 测试底部和顶部链ssDNA供体(如果可能)
本文中的文本和图像改编自IDT网络研讨会:在基因组编辑中使用Cas9 Nickas的优化方法。有关更多信息和存档网络研讨会的链接,点击这里.我们要感谢IDT科学家Mollie Schubert和Shuqi Yan对这篇博文的评论。
Addgene博客上的额外资源188博金宝官网
在Addgene.org上的资源








留言